
2025年8月26日晚(北京时间),复旦大学公共卫生学院、上海市重大传染病和生物安全研究院粟硕教授团队联合中国科学院微生物研究所等多家单位,在国际学术期刊《细胞》(Cell)发表题为“Extensive cross-species transmission of pathogens and antibiotic resistance genes in mammals neglected by public health surveillance(公共卫生监测忽视的哺乳动物中病原体及抗生素耐药基因的广泛跨物种传播)”的研究论文。
哺乳动物不仅包括人类,还涵盖伴侣动物、家畜以及野生哺乳动物等,构成了多样而复杂的宿主体系。这些宿主体内,特别是消化道中,存在着高度复杂且庞大的微生物群落,包括细菌、病毒、真菌等。多数微生物与宿主呈共生或中性关系,在营养代谢、免疫调控和生态稳定性方面发挥关键作用,整体上被称为“微生物组”,构成了宿主重要的内部生态系统。
因此,“哺乳动物微生物组”是指不同哺乳动物宿主体内全部微生物群落的总和。其中部分微生物及其功能元件(如ARGs)能够在不同宿主间发生跨物种传播。所谓“跨物种传播”,通常是指微生物从一种宿主物种传播至另一宿主物种,并在新宿主中适应或持续存在的过程,这一现象对于理解微生物的病原生态学及其公共卫生意义至关重要。
这项研究就像是给这个巨大而隐秘的微生物世界绘制了一张前所未有的“地图”,首次系统解析了大量此前未知的哺乳动物微生物组多样性,并绘制了临床重要ARGs的跨宿主共享网络,拓展了人类对于微生物组成和多样性的认知边界和深度,并为微生物源疾病和抗生素耐药性防控提供重要理论基础。
突破多重技术瓶颈,
构建创新分析框架
哺乳动物体内微生物及其携带的ARGs的跨宿主传播,是潜藏的重大公共卫生风险源。然而,现有研究面临多重技术瓶颈:低丰度微生物难以检测导致潜在病原漏报;大量未报道的微生物物种缺失限制了多样性认知;物种及功能注释精度不足阻碍了明确ARGs与可移动遗传元件(MGEs)的关联;现有宏基因组分析方法分辨率有限(通常仅到物种或属水平),难以精确追踪不同宿主同源菌株或基因的传播,使得解析复杂的跨宿主传播网络极具挑战。
为突破这些关键瓶颈,研究团队构建了两大创新分析框架:
1.交叉多组学高分辨率微生物组解析框架:融合多组学测序、精准微生物基因组重构、新型物种划分策略以及基于基因组比较的同源菌株识别,实现低丰度及新型微生物的精确鉴定,并追踪跨宿主菌株共享事件。
2.ARG与MGE高精度注释框架:通过多数据库交叉验证及ARG-MGE联合分析,实现ARGs和MGEs的精细分类和精准丰度计算,并构建重要ARGs的跨宿主共享网络。
解析7000+未知细菌!全球首次绘制哺乳动物高分辨率微生物图谱
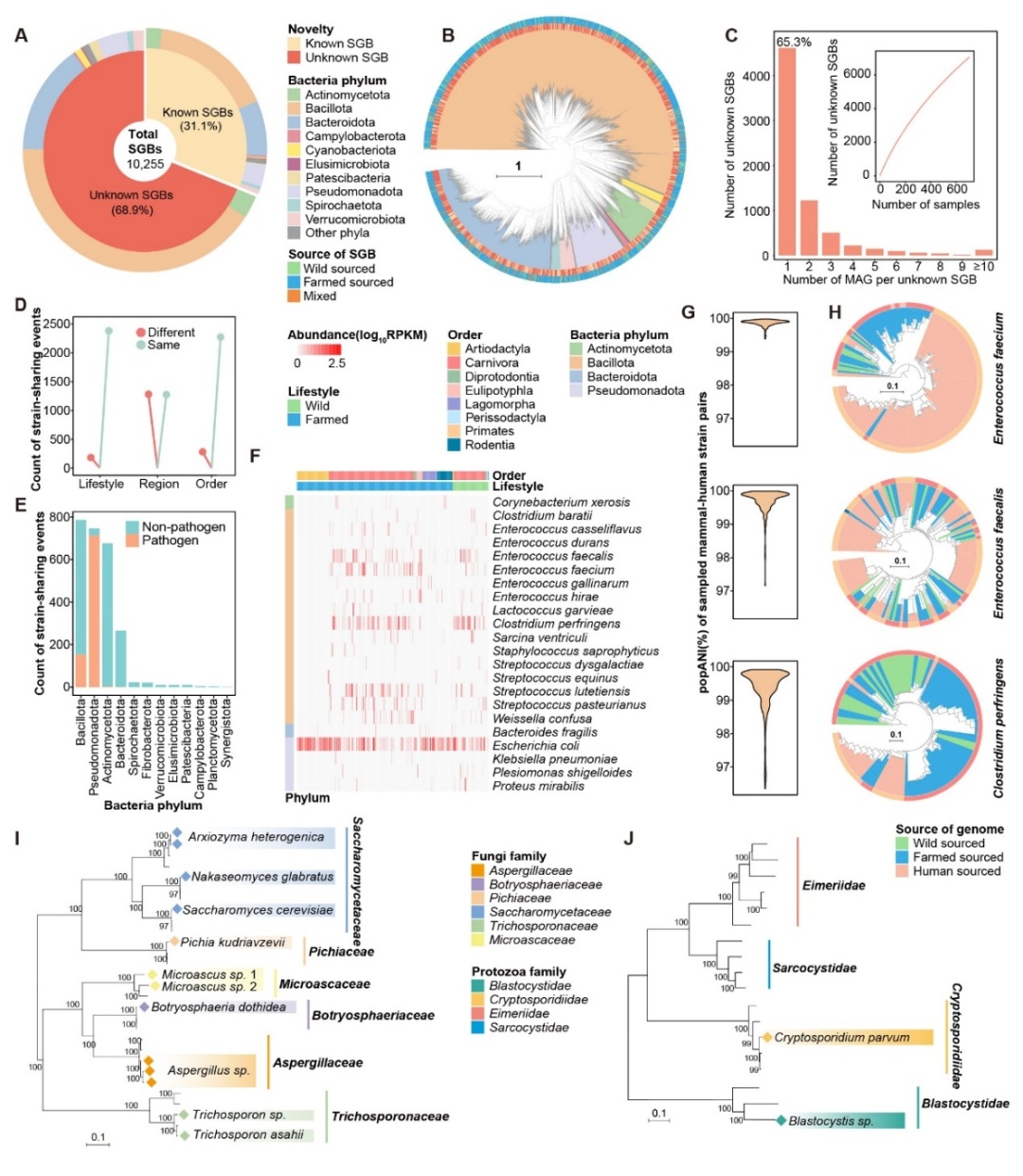
图1:哺乳动物的高分辨率微生物组
基于高分辨率微生物组解析框架,研究团队回收了245个病毒、25,442个细菌、13个真菌和2个寄生虫基因组,极大扩展了现有微生物参考数据库。共鉴定出128种病毒、10,255种细菌、201种真菌和7种寄生虫,其中约70%的细菌物种(超过7,000种)被推测为潜在新物种,揭示了哺乳动物体内丰富的微生物“暗物质”。
多层次群落结构分析显示,微生物群落在相同地理区域、宿主分类、及生活方式下显著相似,表明这些因素是形成与维持群落的关键驱动力。团队利用高分辨率基因组比对与菌株分辨技术,首次系统揭示了哺乳动物微生物组中广泛的跨地理区域、宿主分类及生活方式界限的菌株共享现象,涵盖已知病原菌及非病原菌。例如,常见于导致食物中毒的产气荚膜梭菌,以及易引发尿路感染的肠球菌属菌株,在不同地理区域、宿主分类间表现出高度基因组一致性,提示其存在跨宿主传播潜力。这为深入理解病原菌生态学和精准评估公共卫生风险提供了坚实的科学依据。
构建重要耐药基因跨宿主共享全景网络,揭示潜在公共卫生风险
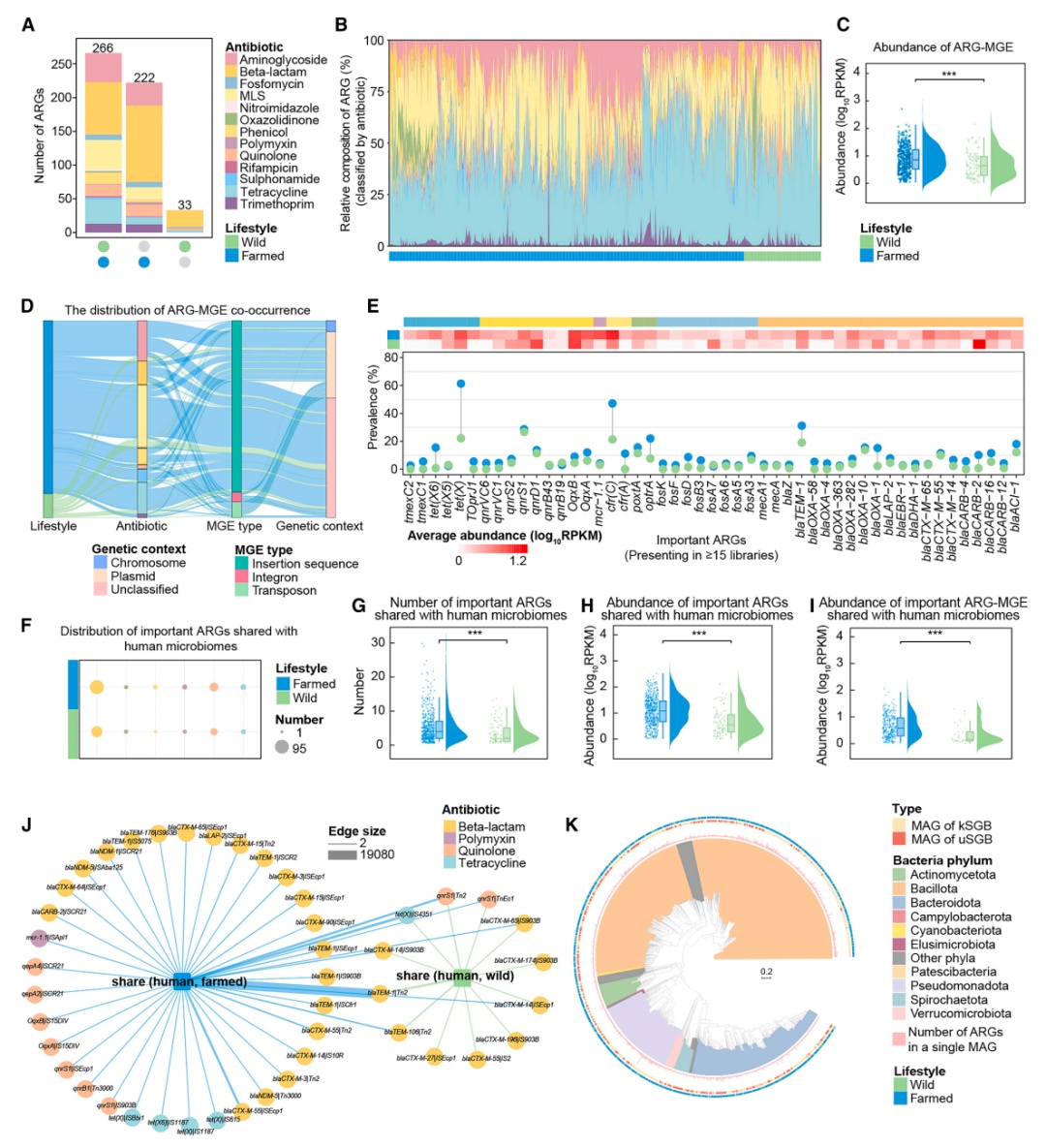
图2:哺乳动物携带的重要ARG的多样性和可转移性
基于ARG与MGE高精度注释框架,在哺乳动物微生物组中鉴定出521种潜在ARGs,涵盖13类抗生素,其中氨基糖苷类、大环内酯-林可酰胺-链霉素类(MLS)、四环素类及β-内酰胺类为主要耐药类别。这些广泛且多样的ARG分布反映了哺乳动物微生物组中长期未被系统评估的耐药潜力。研究中观测到ARGs与MGEs高度共现,其中约五分之四的共现事件由插入序列(IS,一类MGE)介导,提示哺乳动物微生物组可能成为潜在可移动ARGs的储存库。
参照世界卫生组织(WHO)2024年报告标准(“仅授权用于人类”或“最高优先级关键抗生素”),团队共鉴定出270种重要ARG亚型,涉及七类抗生素,其中高频基因如tet(X)、cfr(C)、和qnrS1等在样本库中检出率超过50%,并表现出高丰度,可能会影响多种关键抗生素的疗效。通过与人类微生物组进行高精度比对并构建跨宿主共享网络(基于 aligned length ≥ 500 bp 且 identity ≥ 99% 的ARG序列),团队发现157种重要ARG在哺乳动物与人类微生物组间共享,主要涉及β-内酰胺类和喹诺酮类,提示这些重要ARG存在潜在跨物种交换的风险,需要进一步关注。该框架为系统理解耐药性在生态系统中的传播和评估公共卫生风险提供了创新且强大的工具。
追踪病原与耐药基因,
为精准公共卫生防控奠基
本研究构建的综合多组学高分辨率微生物组解析框架及ARG与MGE高精度注释体系,首次实现了对哺乳动物微生物组中低丰度及新型微生物的精确鉴定,以及对病原菌株和临床重要ARGs跨宿主传播路径的高精度追踪。
该框架深度融合微生物群落生态学、进化生物学、系统生物学和比较基因组学等,整合了自有高深度多组学测序数据及公共数据库资源,构建起跨尺度、多维度的微生物组与抗性组解析体系,实现复杂的微生物和ARGs跨宿主网络的高精度可追踪分析。未来,这一基于多学科前沿技术交叉的强大框架可应用于探索人类对微生物的认知边界和深度,系统监测新发病原体、预测重要耐药性扩散趋势,为制定精准、高效的公共卫生防控策略提供核心技术支撑。
复旦大学公共卫生学院粟硕教授为论文通讯作者,在读博士研究生石宇旗等为论文共同第一作者。本研究得到了上海市优秀学术带头人项目和教育部U40项目等资助。
原文链接:
https://doi.org/10.1016/j.cell.2025.08.016


来 源
公共卫生学院、医学宣传部
文 字
石宇旗、孙芯芸
图 片
受访者提供、黄奕宁
责 编
殷梦昊
编 辑
黄奕宁

▼更多复旦新闻,敬请留意复旦大学官方网站。